Laboratorio di Biologia Evolutiva
Scienze – Edificio 3 – 2° piano
(Responsabile del laboratorio: Prof. Vincenzo Caputo Barucchi)
 |
Nel Laboratorio di Biologia Evolutiva (EvoLab_DiSVA) si studia la diversità genetica animale mediante l’utilizzo di marcatori molecolari nucleari e mitocondriali. Si eseguono attività di campionamento, estrazione del DNA, amplificazione tramite PCR (Polymerase Chain Reaction), genotipizzazione tramite microsatelliti, analisi bioinformatiche di dati da sequenziamento Sanger e NGS (Next Generation Sequencing). Le analisi riguardano Vertebrati marini e d’acqua dolce di particolare interesse commerciale e conservazionistico nell’area mediterranea e vengono effettuate al fine di chiarirne lo status di conservazione e promuoverne una corretta gestione. Alle analisi su campioni moderni è spesso affiancata quella di campioni storici e antichi con lo scopo di evidenziare direttamente cambiamenti nel tempo della diversità genetica di popolazioni naturali causati dall’impatto antropico e/o dai cambiamenti climatici del passato. |  |
Attività di ricerca
- GENETICA E GENOMICA DELLA CONSERVAZIONE DELLA TROTA MEDITERRANEA
-
La trota fario (Salmo trutta complex) è una specie nativa dell'Europa, dell'Asia occidentale e del bacino del Mediterraneo. Questo complesso di specie comprende forme geograficamente distinte, che possono però incrociarsi tra loro, e che mostrano un’elevata plasticità fenotipica, oltre ad una straordinaria diversità genetica ed ecologica. La sopravvivenza delle popolazioni dell’area mediterranea è però messa fortemente a rischio dalla distruzione degli habitat fluviali e dall'ibridazione con trote alloctone introdotte a sostegno della pesca ricreativa. Da oltre vent’anni, EvoLab_DiSVA studia l'evoluzione e la diversità genetica di S. trutta al fine di fornire utili indicazioni per la conservazione e corretta gestione delle popolazioni italiane a serio rischio di estinzione. Obiettivi in parte raggiunti grazie al coordinamento scientifico di un progetto europeo (LIFE12 NAT/IT/000940 “Trout Population Recovery in Central Italy”) e attraverso numerose attività di collaborazione scientifica con enti pubblici italiani.



- ANALISI STOCK GENETICI DI SPECIE ITTICHE DI INTERESSE COMMERCIALE
-
Molte specie di Pesci Teleostei hanno subito drastici crolli demografici a causa dell’eccessiva ed incontrollata attività di pesca, fenomeno noto con il termine di “overfishing”. La riduzione in taglia delle popolazioni naturali può provocare la perdita di variabilità genetica (collo di bottiglia o “bottleneck”), determinando una riduzione del potenziale riproduttivo ed evolutivo delle specie. EvoLab_DiSVA svolge da anni ricerche genetiche su specie ittiche di interesse commerciale, sia piccoli (e.g. acciuga Engraulis encrasicolus e sardina Sardina pilchardus) che grandi pelagici (e.g. pesce spada Xiphias gladius), allo scopo di valutare la struttura genetica delle popolazioni ed evidenziare significative correlazioni tra fluttuazioni demografiche e della variabilità genetica. Lo svolgimento di queste attività di monitoraggio è importante per individuare eventuali unità gestionali separate all’interno delle specie e per svelare la presenza e gli effetti di fenomeni di “overfishing”.



- INDAGINI GENETICHE E GENOMICHE SU CETACEI
-
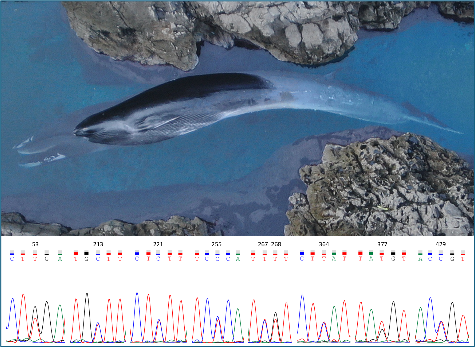
I Cetacei svolgono un ruolo ecologico cruciale negli ecosistemi marini, sia come predatori apicali sia come consumatori di plancton, e rivestono un particolare interesse conservazionistico in quanto minacciati dalle attività antropiche (e.g. catture accidentali, collisioni con navi, inquinamento chimico e acustico) e dai cambiamenti climatici. EvoLab_DiSVA esegue analisi genetiche e genomiche allo scopo di monitorare lo status conservazionistico delle popolazioni di Cetacei residenti nel Mar Mediterraneo e di svelare identità e provenienza di individui appartenenti a specie che solo occasionalmente visitano il bacino. Inoltre, le analisi svolte permettono di evidenziare eventuali fenomeni di ibridazione interspecifica che, come ipotizzato per la Balenottera azzurra (Balaenoptera musculus), potrebbero portare ad un declino della diversità genetica e ad un conseguente aumento del rischio di estinzione.
- DNA STORICO E ANTICO
-
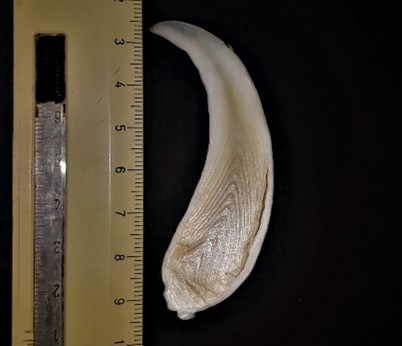
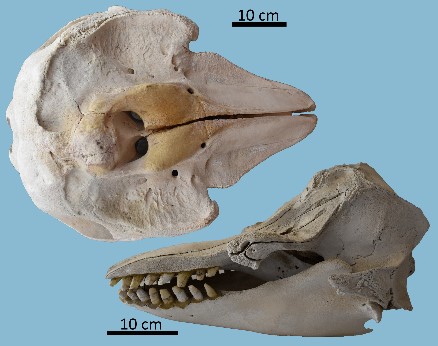
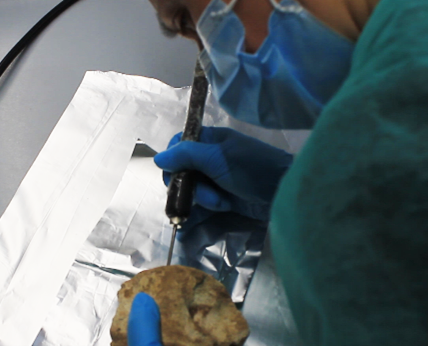
Il gruppo di ricerca di EvoLab_DiSVA si avvale di un laboratorio dedicato all’analisi del DNA da campioni storici e antichi (e.g. esemplari tassidermizzati e in formalina, ossa e denti da collezioni museali e scavi archeologici). La separazione fisica dal laboratorio principale, in cui si eseguono le analisi su campioni moderni, permette il controllo della contaminazione da DNA esogeno a cui è spesso soggetto il DNA storico/antico, essendo degradato in corti frammenti e presente in ridotta quantità nei campioni in esame. L’analisi di campioni da collezioni museali è particolarmente importante per lo studio genetico di specie rare e difficili da campionare in natura. Inoltre, il confronto di dati genetici ottenuti da campioni storici/antichi e moderni permette di evidenziare fluttuazioni della variabilità genetica delle specie nel tempo ed identificarne le possibili cause (attività antropica vs. cambiamenti ambientali).


- Progetti di ricerca finanziati
-
2024-2025: Convenzione di ricerca con la Città Metropolitana di Torino nell’ambito di un progetto transfrontaliero con la Francia ( Interreg VI ALCOTRA 21-27 – Progetto N. 20125 “Ge.co - Gestione Ecologica dei Conflitti” WP 3.1) sul monitoraggio genetico della trota mediterranea al confine italo-francese.
2023-2024: Convenzione di ricerca con la Regione Marche per il monitoraggio genetico della trota mediterranea nel territorio regionale.
2020-2022: Progetto di tutela e gestione dei rifugi genetici della trota mediterranea in Sardegna - registrazione n. 296 del 01.09.2020.
2020-2021: Rinnovo della intesa con la Città Metropolitana di Torino per il monitoraggio genetico delle popolazioni del genere Salmo delle Alpi occidentali.
2019: Affidamento da parte dell’Ente Parco Nazionale del Gran Paradiso di una prestazione di servizio per analisi genetiche su campioni di trota marmorata.
2018-2020: Intesa con la Città Metropolitana di Torino per il monitoraggio genetico delle popolazioni del genere Salmo delle Alpi occidentali.
2018: Intesa con la Provincia di Cuneo per analisi genetiche di campioni di trota fario delle Alpi occidentali.
2017: Convenzione col “Museo delle Civiltà” di Roma su “DNA antico, evoluzione di Salmonidi mediterranei, paleoclimatologia, paleontologia del Quaternario, archeozoologia e pedologia”.
2017: Affidamento da parte dell’Ente di gestione delle aree protette del Monviso di una prestazione di servizio relativa all'analisi genetica e valutazione dei risultati di campioni di Salmo trutta raccolti in parco e in aree limitrofe di controllo nel contesto delle Alpi sud occidentali.
2016: Progetto strategico di Ateneo “Integrating approaches (ancient DNA, climate stratigraphy, pedology) to open a window on the past: the case of prehistoric trout in the mediterranean basin during the great climatic change across Pleistocene and Holocene”.
2016-2020: Accordo di collaborazione tra la regione Umbria, il Dipartimento di Chimica, Biologia e Biotecnologie, dell’Università degli Studi di Perugia e il Dipartimento di Scienze della Vita e dell’Ambiente dell’Università Politecnica delle Marche per l’approfondimento di conoscenze relative alla fauna ittica del bacino del fiume Tevere e per la ricerca, selezione e produzione di trote mediterranee da ripopolamento.
2016: Convenzione per la realizzazione del progetto “redazione di una carta ittica delle acque dolci della Sardegna con particolare riferimento ai siti di popolamento della forma geneticamente pura della trota sarda (Salmo cettii ex Salmo trutta macrostigma), specie autoctona della Sardegna a grave rischio di estinzione".
2013-2017: LIFE-NATURA 2012, PROPOSAL LIFE12 NAT/IT/000940 "Trout Population Recovery in Central Italy".
2013: Convenzione con il Parco Nazionale dei Monti Sibillini, “misure urgenti per la salvaguardia del gambero di fiume (Austropotamobius pallipes)”.
2013: Convenzione di ricerca con la Provincia di Terni: “selezione, recupero e conservazione del ceppo mediterraneo autoctono di trota fario (Salmo ghigii pomini, 1941) nel bacino del fiume Nera”.
2012: Progetto RITMARE, responsabile di unità di ricerca (SP2_WP1_AZ4_UO02, la struttura genetica degli stock di acciuga, Engraulis sp., nel Mare Adriatico).
2011: PRIN2009 “La determinazione del sesso nei rettili squamati: aspetti cromosomici e molecolari”.
2010: Convenzione con l’associazione “Sistema museale della provincia di Ancona”, finalizzata alla collaborazione per lo sviluppo della ricerca dal titolo: “biodiversità delle Marche documentata dai reperti zoologici del Museo di Scienze Naturali “Luigi Paolucci” di Offagna.
2009: Convenzione con la Provincia di Pesaro e Urbino per la ricerca condotta nell’ambito del progetto dal titolo “screening genetico propedeutico ad attività di “supportive breeding” in popolazioni di trota fario (Salmo trutta l. complex) nei torrenti nella provincia di Pesaro Urbino”.
2008: convenzione con la Provincia di Ancona per la ricerca condotta nell’ambito del progetto dal titolo “screening genetico su popolazioni di trota fario (Salmo trutta l. 1758) nella provincia di Ancona”.
2006: Convenzione di ricerca con la società “ASTERIA srl” sul progetto dal titolo: “TRUENTUM FLUMEN, azioni per la conoscenza e la biodiversità del fiume Tronto”.
- Strumentazione
-
Agitatore magnetico riscaldante ArgoLab M2-A
Agitatore orbitale IKA KS 260 basic
Agitatore vortex ArgoLab Mix
Autoclave ASAL 760
Apparati per elettroforesi verticale Bio-Rad Sequi-Gen cell e Biometra Model S2
Bagno termostatico Grant TC120
Bagnomaria Dubnoff a scuotimento orizzontale ASAL 740
Bilancia analitica Radwag AS160.2
Cappe (chimica e a flusso laminare)
Cappe di decontaminazione AURA PCR
Celle elettroforetiche orizzontali Mini-Sub Cell GT Cell e Wide Mini-Sub Cell GT Cell
Centrifughe da banco Eppendorf MiniSpin, Eppendorf Centrifuge 5424R e ALC Centrifuge 4235A
Estrattore automatico di acidi nucleici Magcore HF16
Fluorimetro Promega Quantus
Generatori Bio-Rad powerpack 3000, Bio-Rad Powerpack HC, Bio-Rad Powerpack HV
Microscopi a epifluorescenza
Microtomo
Termomiscelatore Eppendorf ThermoMixer C (in dotazione: Coperchio riscaldato Eppendorf Thermotop e blocchi termici Eppendorf Smartblocks 2.0-1.5-0.2 ml)
Termociclatori Bio-Rad T100
Transilluminatore Moonlight con sistema di acquisizione e analisi di immagini
Staff
|
RESPONSABILE DEL LABORATORIO Prof. Vincenzo Caputo Barucchi https://orcid.org/0000-0002-4427-3129 v.caputo@staff.univpm.it Laureato in Scienze naturali e dottore di ricerca in Biologia evoluzionistica, è attualmente Professore Ordinario di Anatomia Comparata presso il Dipartimento di Scienze della Vita e dell’Ambiente, dove insegna “Anatomia comparata” (Laurea triennale in Scienze Biologiche) e “Biologia evolutiva dei Vertebrati marini” (Laurea magistrale in Biologia marina). Ha svolto ricerche faunistiche nell’area del Bacino mediterraneo (Europa meridionale e Nordafrica) e ha partecipato alla XIV e XVII Spedizione Italiana in Antartide. Nel corso di queste indagini ha avuto l’opportunità di scoprire alcune specie di vertebrati nuove per la Scienza. I suoi attuali interessi di ricerca riguardano la biologia della conservazione di vertebrati marini e di acqua dolce e lo studio del DNA antico ottenuto da campioni museali e archeologici. |
 |
|
Dott. Andrea Splendiani Laurea Magistrale in “Biologia Marina” e Dottorato di Ricerca in “Biologia ed Ecologia Marina” presso il Dipartimento di Scienze della Vita e dell’Ambiente (UNIVPM). Esperto nella genotipizzazione di specie e popolazioni di vertebrati (con particolare riguardo ai pesci teleostei) attraverso marcatori mitocondriali e nucleari e nell’elaborazione dei dati attraverso l’utilizzo di specifici pacchetti statistici. Autore e coautore di numerose pubblicazioni internazionali peer-reviewed; membro del Consiglio Direttivo dell’Associazione Italiana Ittiologi Acque Dolci; Editor Associato della rivista The European Zoological Journal. |
 |
|
Dott.ssa Tatiana Fioravanti Laurea Magistrale in “Biologia Marina” e Dottorato di Ricerca in “Biologia ed Ecologia Marina” presso il Dipartimento di Scienze della Vita e dell’Ambiente (UNIVPM). Esperta in genetica di popolazione di vertebrati marini e d’acqua dolce, e specializzata nell’analisi del DNA estratto da campioni antichi e storici. Autrice e coautrice di numerose pubblicazioni internazionali peer-reviewed. |
 |
|
Dott.ssa Lucrezia Latini Attualmente iscritta al XXXIX ciclo del Dottorato di Ricerca in “Biologia ed Ecologia Marina”. Laurea Magistrale in “Biologia Marina” con focus della ricerca sui Cetacei sia dal punto di vista genetico che scheletrocronologico per la determinazione dell’età. |
 |

